👋 Hi
这个博客主要用于记录我日常的学习、工作和想法。
如果有什么问题,可以通过邮箱: lideanlda@foxmail.com与我联系。

Signac是一个可以处理scATAC-seq、scCUT&Tag和scNTT-seq等检测单细胞水平染色质状态的R包,其主要步骤包括:
Create object
QC
Normalization
Dimension Reduction and Clustering
Inference of gene activity
Identify differential peaks
Plotting genomic regions
CytoTRACE2是 Newman Lab 开发的用于预测scRNA-seq数据中细胞干性和发育潜能的计算方法。
CytoTRACE2本质是一种深度学习框架,该模型在涵盖 28 种组织类型和整个发育范围的人类和小鼠 scRNA-seq 数据集的训练和验证(总共31个数据)。利用该模型,CytoTRACE2可以将细胞分类为终末分化(differentiated: 0)和全能细胞(totipotent: 1).
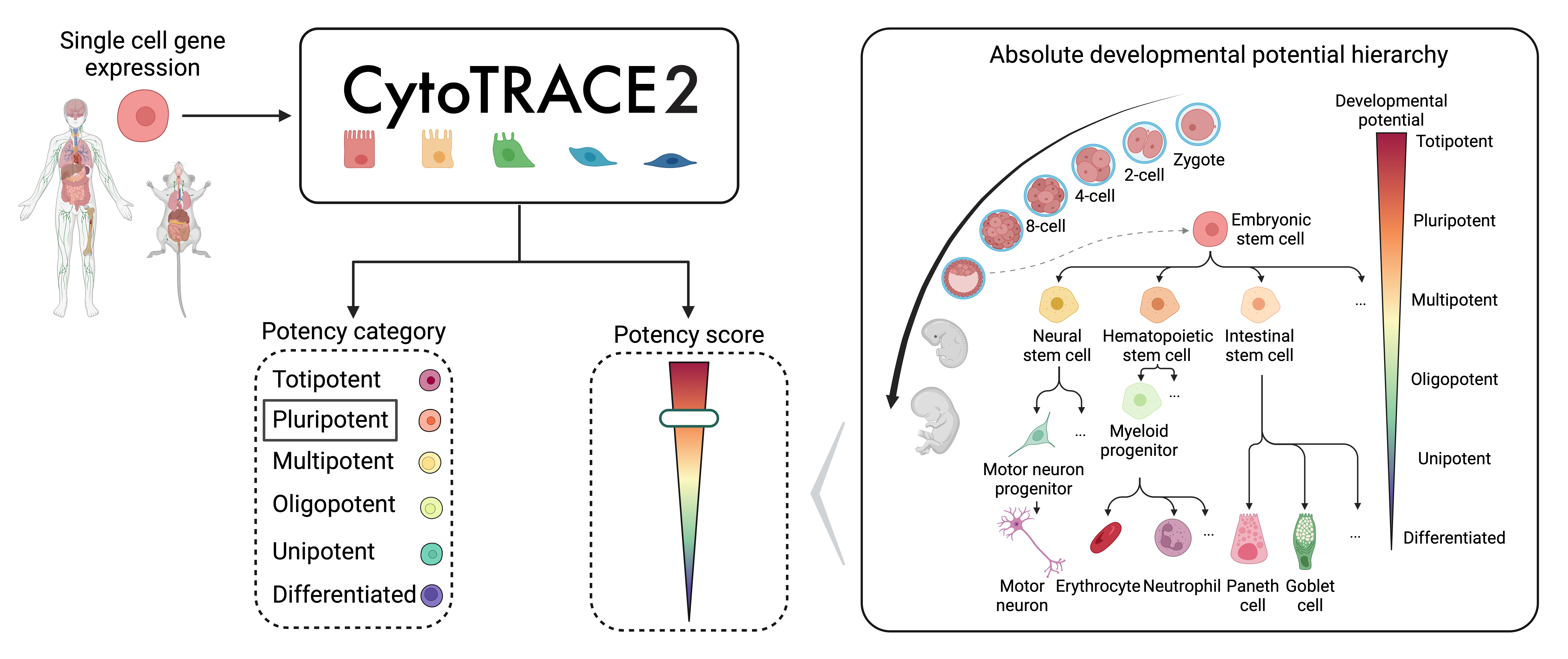
本文简单介绍CytoTRACE2 R包的基本用法,具体原理可参考他们目前的预印本 https://doi.org/10.1101/2024.03.19.585637
最近想将单细胞数据从Seurat转还到H5AD格式,但发现Seurat V5直接转换会报错。这里记录一下解决的方案。
最近开发了一个R包,enONE(https://github.com/thereallda/enONE),可以用于NAD-RNA-seq或者其他代谢物修饰RNA的数据分析中。相关工作也发表了,欢迎大家使用并提出意见 :)

Epitranscriptome analysis of NAD-capped RNA by spike-in-based
normalization and prediction of chronological age
(https://doi.org/10.1016/j.isci.2023.108558)