最近在看Construction of a human cell landscape at single-cell level 这篇文章。其中,作者构建出了多组织的人类单细胞图谱。注意到作者使用了Microwell-seq这一种scRNA-seq,因此特意了解该技术,来跟大家分享一下我对这项技术的理解。
Microwell是什么?
Microwell-seq 是浙江大学郭国冀等人在2018 Cell中提出的一种scRNA-seq方法。
Microwell-seq的特点是使用一种特殊的琼脂糖微孔板,即Microwell作为单细胞捕获的平台。
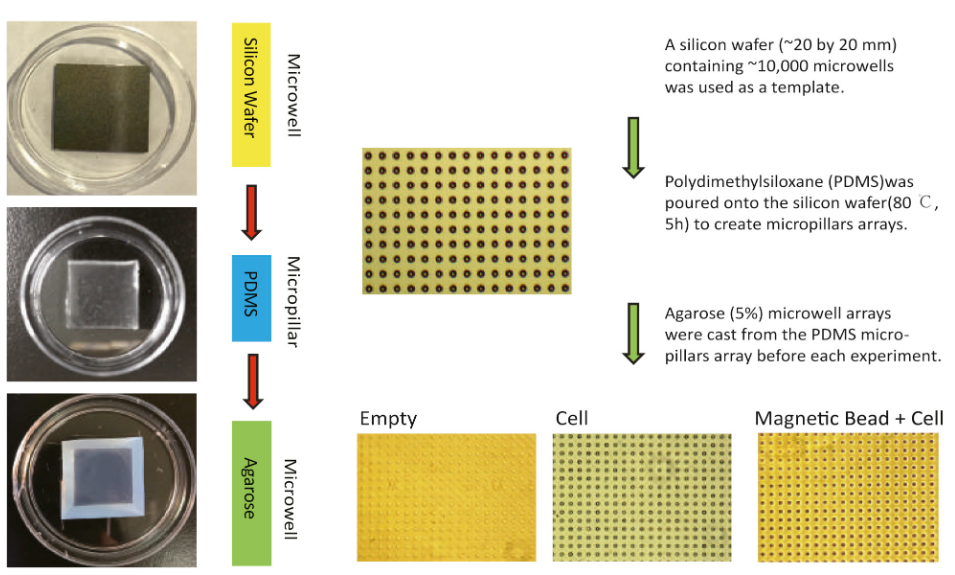
Microwell的制备如上图所示,其本质是由刻有约10万个小孔的硅片作为载体,再在其上包裹上PDMS和Agarose而成。从图中可以看到,单细胞加样到Microwell后会落入芯片的小孔中,一个小孔只能容纳单个细胞与一个磁珠(其protocol指出一般只有10%的小孔既有细胞又有beads)。图中有黑点的孔就是有细胞loading上的,每一个小孔相当于一个独立的反应腔,之后的细胞裂解,磁珠吸附操作都会在此完成。如此就保证了一个磁珠上的RNA只来源于一个细胞。
建库步骤
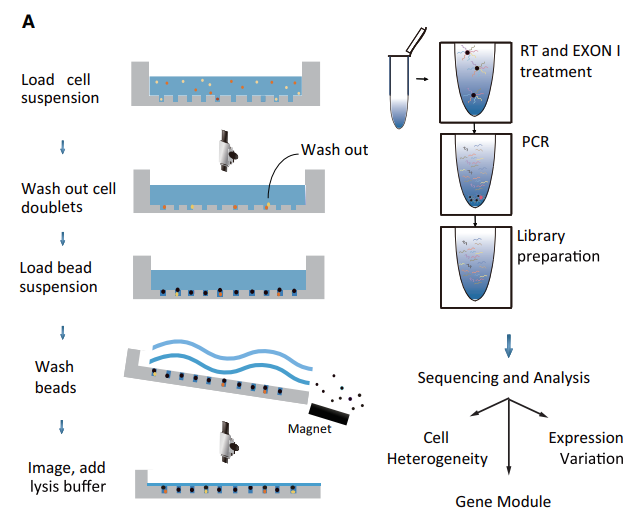
Microwell-seq 建库流程如上图所示,基本包括:
- 分离单细胞悬液,并加上Microwell,进行单细胞捕获。通过显微镜检查细胞落入小孔的比例
- 洗脱未加进小孔的细胞,或者一个孔中有两个细胞的doublet
- 加入磁珠
- 洗掉悬浮的磁珠
- 镜检后,加入裂解液进行细胞裂解。细胞裂解后,RNA会吸附到磁珠的primer上。
- 将磁珠转移到EP管中进行reverse transcription合成cDNA,并使用EXON I 切割。
- 随后利用PCR扩增,构建文库。文库构建完成后便可上机测序(可使用Illumina测序仪)
Cell barcoding
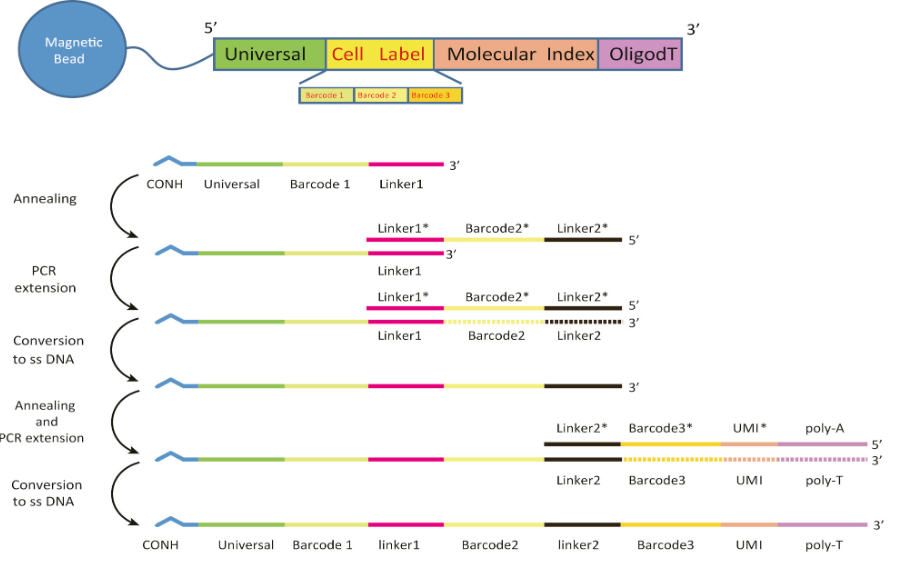
Microwell-seq的barcode也是利用一种UMI的方法区分不同细胞来源的mRNA。对mRNA的富集是通过oligo(dT)实现的,所以会有3’ bias
优缺点

优点
- 捕获单细胞的硅片可以反复利用,以此节约设备的成本。
- 细胞通量在5k以上,比FACS方法的细胞通量高。
- 单个文库构建成本较低,
- 在细胞分选的过程中加入镜检质控,提高单细胞分选质量,
缺点
- 利用oligo(dT)捕获mRNA,存在3’ bias
- 细胞分离过程使用镜检,虽然提高了单细胞分选质量,但也增加了操作过程。
一点看法
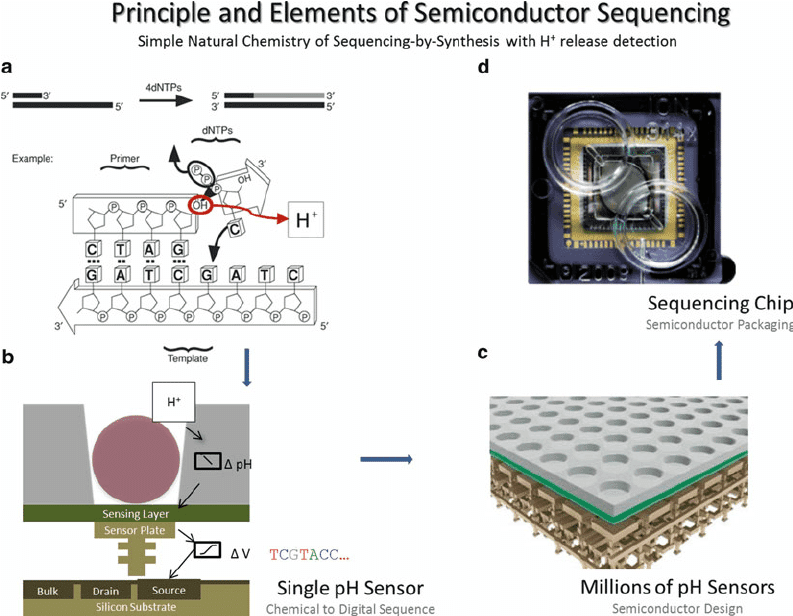
Microwell-seq 是由华人科学家独立开发的一种单细胞测序技术,有高通量、低成本和测序质量高的特点。Microwell给我的感觉与Iontorrent的sequencing chips很相像。Iontorrent的chips是通过将beads加到一个个小孔中测序,以此辨别每个beads上的测序信号。而Microwell则是将单细胞和beads加入小孔中进行反应,本质上小孔都是提供一种独立反应腔的功能,保证各个小孔的信号不会互相干扰。这也是单细胞分离的要点,即准确地标记出RNA的细胞来源。
以上就是我对Microwell-seq一点粗略的见解。
完。
Reference: