Smart-seq是由路德维格癌症研究所的 Rickard Sandberg实验室所开发的一套在全转录组范围进行单细胞RNA测序 (scRNA-seq) 的方法。Smart-seq因为以全长mRNA建库,所以对转录本的测序覆盖度也有所上升。Smart-seq2是由Picelli等人从Smart-seq中改良而来(Picelli et al., 2013) (Picelli et al., 2014)。本文将从Smart-seq2的原理、优点和缺点来为大家介绍这项scRNA-seq技术。
Smart-seq2建库原理
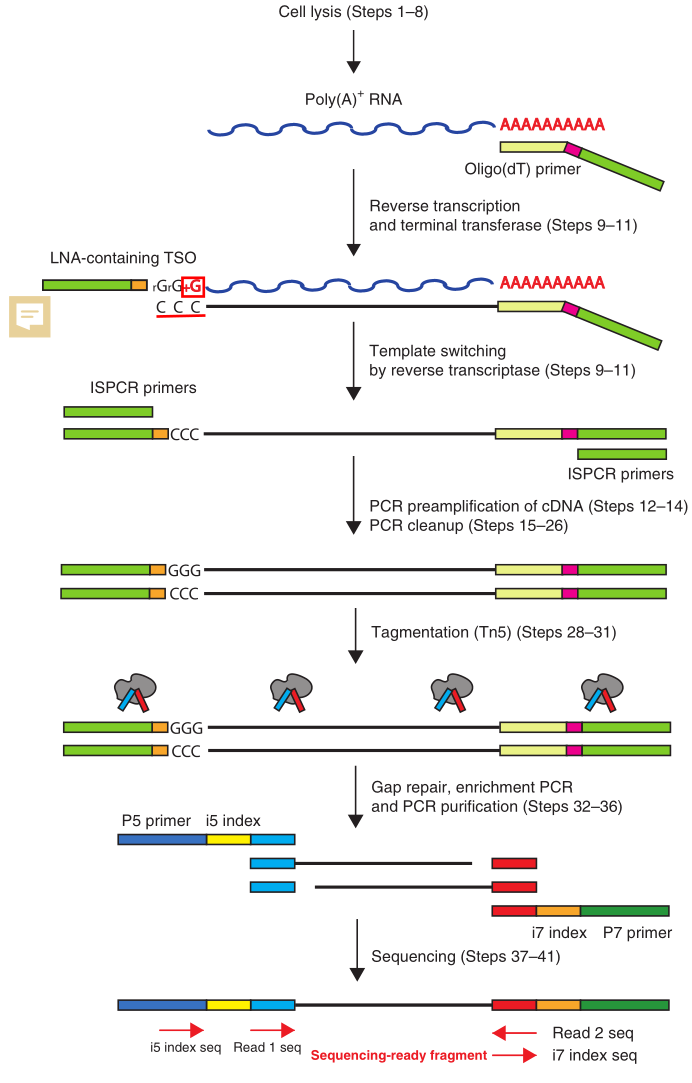
Smart-seq2的建库原理如下:
- 单细胞分选:Smart-seq使用流式细胞仪进行细胞分选,一次最大分选细胞通量为96个。
细胞裂解:在细胞裂解液中进行细胞裂解。
反转录( 一链合成 ):使用Oligo(dT) primer 对带有polyA尾的RNA( 主要是mRNA )进行反转录。由于使用了鼠源的反转录酶( Moloney Murine Leukemia Virus )进行反转录,所以会在cDNA链3’端加上三个C
模板置换( 二链合成 ):该步使用TSO ( template-switching oligo )引物合成了cDNA的二链,从而置换了与一链cDNA互补的RNA。要注意的是TSO 3’端有三个G能与一链3’端的三个C互补,而最末端的+G是一个修饰过的G,能增加TSO的热稳定性,以及其与一链cDNA游离的3’端的互补的能力。TSO 还带有PCR所需的引物(图中绿色的部分)。
PCR扩增:该步进行轻度的cDNA富集,将cDNA扩增至ng级即可。
标记:利用改造后的高活性Tn5转座酶对DNA进行打断的同时将接头添加到cDNA的两端。标记完成后的DNA片段通常在200-600bp
PCR富集&上级测序:在进行最后一次PCR扩增后,即可上机测序。
Smart-seq2使用的测序仪为一般的Illumina 测序仪,因其文库最后都与一般的bulk RNA-seq相差无几。
优缺点
优点:
- 以全长mRNA建库,对转录本覆盖度很高,能够知道每个外显子的表达量,甚至可以获取转录本剪接的信息
- (号称)50pg RNA起始建库
- 提高了可mapping reads的比例,因为较少存在Drop-seq那种空beads的无用reads的情况
- mRNA sequence does not have to be known(?我觉得没有几种大规模测序需要提前知道序列吧)
缺点:
- 非链特异。无法区分正反链reads
- No early multiplexing(我的理解是建库前期没有加入UMI所以对单个细胞的分辨能力很低)
- 转录本长度偏好,对于大于4kb的转录本建库效率较低
- 对于高丰度转录本的PCR偏好。在PCR过程中必然有丰度偏好性,这也是现在许多使用了PCR进行文库扩增的高通量测序protocol无法避免的”硬伤“。
- 潜在的链侵入偏好。Tn5转座酶可能会偏好对某种特征的链进行更高效的链侵入。
- 使用流式细胞仪进行细胞分选,效率较低,一次测序细胞通量不高。
完。
Ref:
- https://www.illumina.com/science/sequencing-method-explorer/kits-and-arrays/smart-seq2.html
- Full-length RNA-seq from single cells using Smart-seq2
- Comparative Analysis of Single-Cell RNA Sequencing Methods
- http://cn.fluidigm.com/applications/single-cell-analysis