整理ChIP-seq / CUT & Tag 分析时用到的工具。本文只对使用的工具用法进行简单介绍。
bedtools-intersect
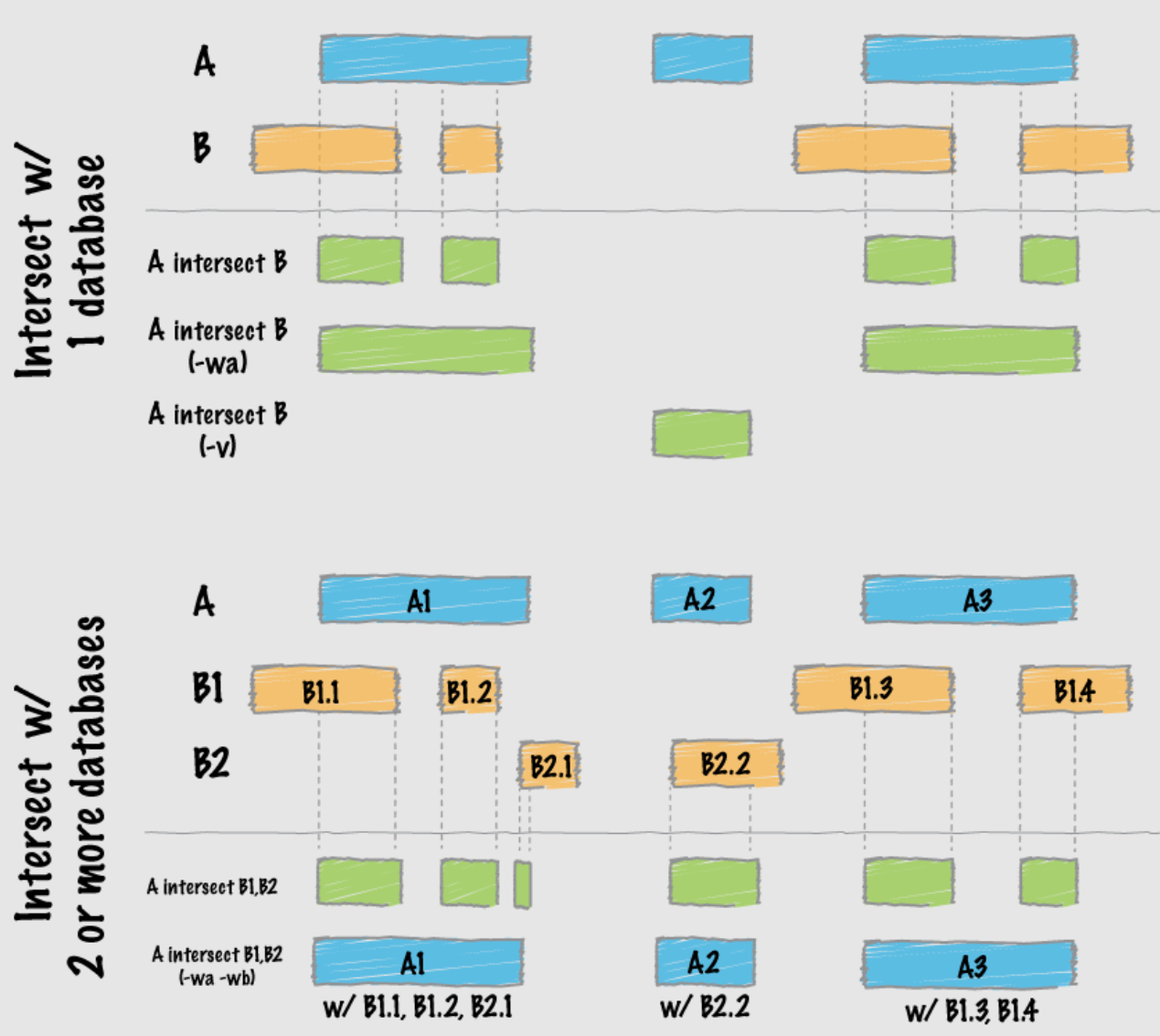
当我们想判断两个基因组区域,例如两个peaks是否重叠时,可以使用bedtools intersect解决这种问题。
整理ChIP-seq / CUT & Tag 分析时用到的工具。本文只对使用的工具用法进行简单介绍。

Bowtie 2是常用的基因组比对软件。其原理在此展开,有兴趣的同学可以参阅其官方文档以及其发表的文章(https://doi.org/10.1038/nmeth.1923)。下面简单介绍Bowtie 2 Index和比对的命令及个人常用参数。
整理ChIP-seq / CUT & Tag 分析时用到的工具。本文只对使用的工具用法进行简单介绍。
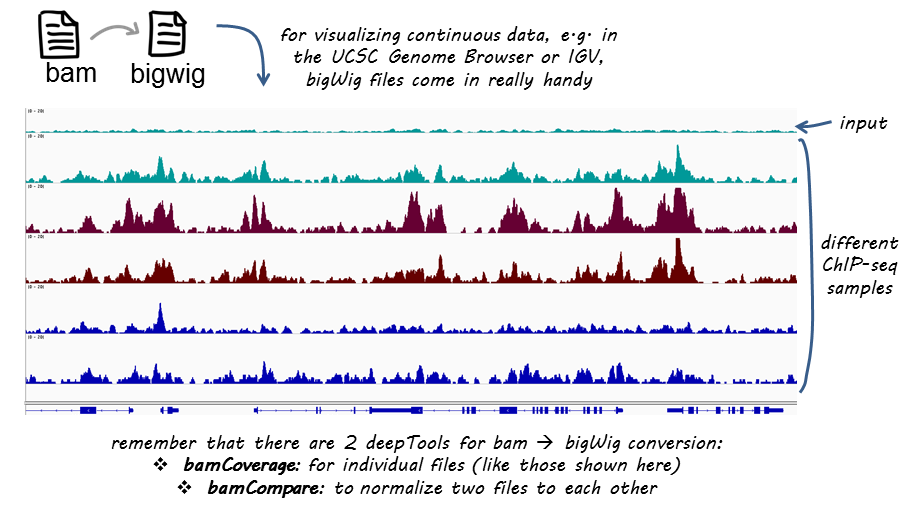
deeptools 提供两个bam转换为bw的命令,分别是bamCoverage和bamCompare。两者的区别在于bamCoverage是对单个bam文件进行转换,而bamCompare接受两个bam文件,生成两者信号比值的bw文件。
整理ChIP-seq / CUT & Tag 分析时用到的工具。本文只对使用的工具用法进行简单介绍。
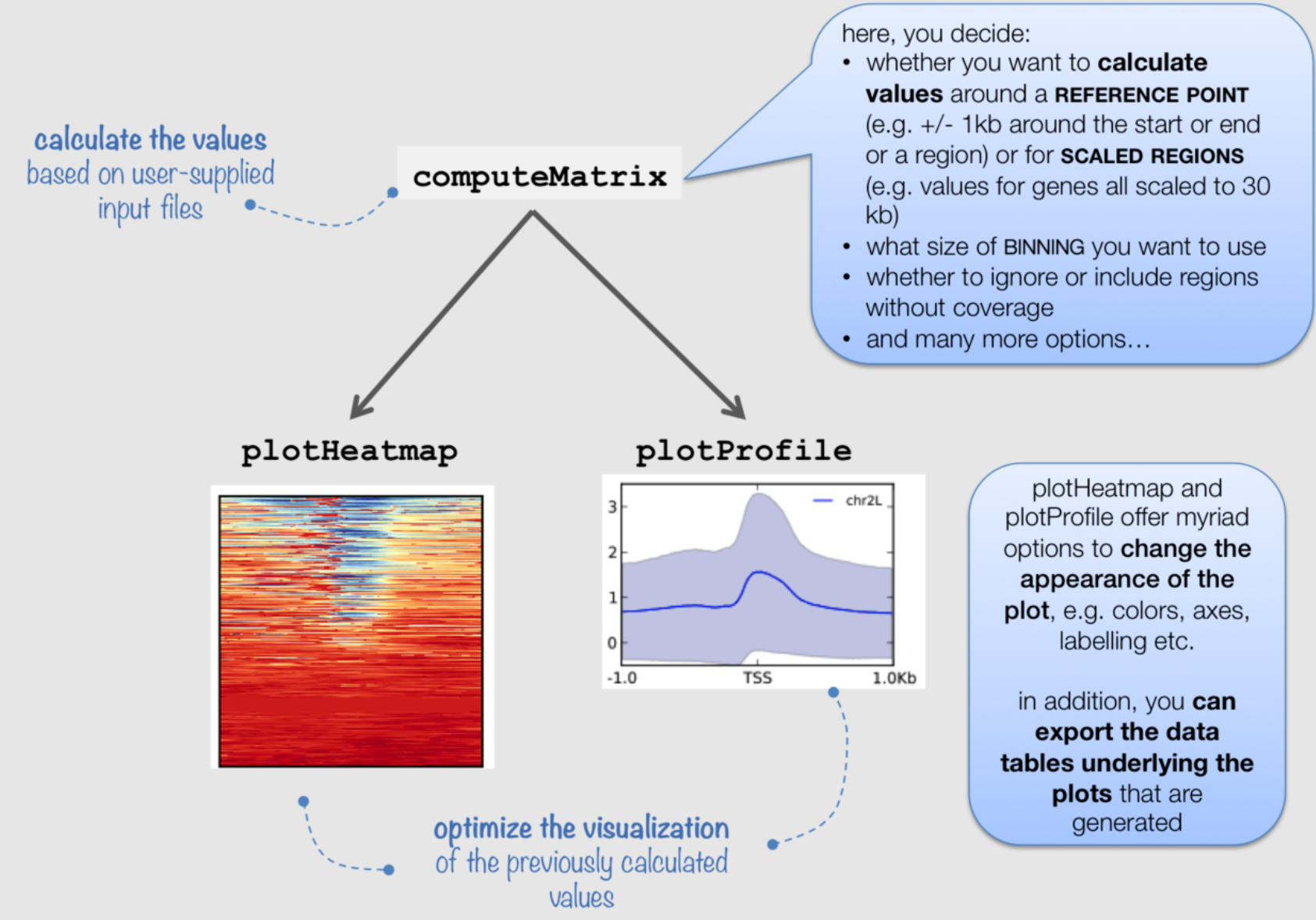
deeptools 提供了computeMatrix命令以计算特定基因组区域的ChIP-seq信号,并通过plotHeatmap和plotProfile函数分别产生ChIP-seq信号热图与谱图。
整理ChIP-seq / CUT & Tag 分析时用到的工具。本文只对使用的工具用法进行简单介绍。
当我们需要评估ChIP-seq类测序数据的相关性时,deeptools 是一个可行且方便的工具。它提供了一系列方便的命令对高通量测序数据进行分析。本文先集中介绍deeptools中计算ChIP-seq样本间相关性所用到的命令,其余的命令有机会再一一介绍。
最近在处理服务器上的R,顺便就想往服务器上装一个RStudio。因此就有了本文记录一下安装及配置的过程。我们的服务器是CentOS 7系统,其他发行版安装过程其实也大同小异。